近日,微软研究院发布了“BiomedParse”模型,该模型为生物医学图像分析提供了一个全面、一体化的解决方案。BiomedParse的出现极大地改变了传统生物医学图像处理的方式,使得对象识别、检测和分割这三个任务在同一个框架中统一实现,从而实现更加智能化和整体化的图像分析。
一、背景与挑战
在现代医学,尤其是癌症诊断和免疫治疗等高度精确的医疗操作中,医学图像的每一个细节都至关重要。病理学家和放射科医生依靠这些图像来识别肿瘤、了解其边界,以及分析其与周围细胞的相互作用。这些分析任务对于肿瘤的诊断、治疗方案的制定和病情监测具有至关重要的作用。当前的生物医学图像分析工具往往将对象识别、检测和分割这三项任务分开处理,导致了不同任务之间缺乏协调和互补性,从而限制了对图像的整体性分析。
以目前的主流工具为例,如MedSAM和SAM,这些工具主要关注对象的分割,但没有涉及对象的识别和检测。因此,这些工具的应用在面对复杂医疗场景时显得力不从心,无法有效地进行全局图像的解析和理解。而生物医学中的许多关键步骤,如肿瘤检测、边界定位和多种结构的识别,要求这些任务能够相辅相成地进行分析,因此传统工具的分离式方法具有明显的局限性。
二、BiomedParse的创新性解决方案
BiomedParse提出了一种创新性的解决方案,通过将对象识别、检测和分割统一到一个综合性框架中,极大地增强了生物医学图像的解析能力。用户只需要通过简单的自然语言描述,BiomedParse即可理解用户的需求并自动进行对象的识别和分割,消除了传统方法中依赖手动设置边界框的繁琐步骤。这种方式提高了生物医学图像分析的效率和精度,使得整个过程更加自然和人性化。
1. 数据集的构建与预训练
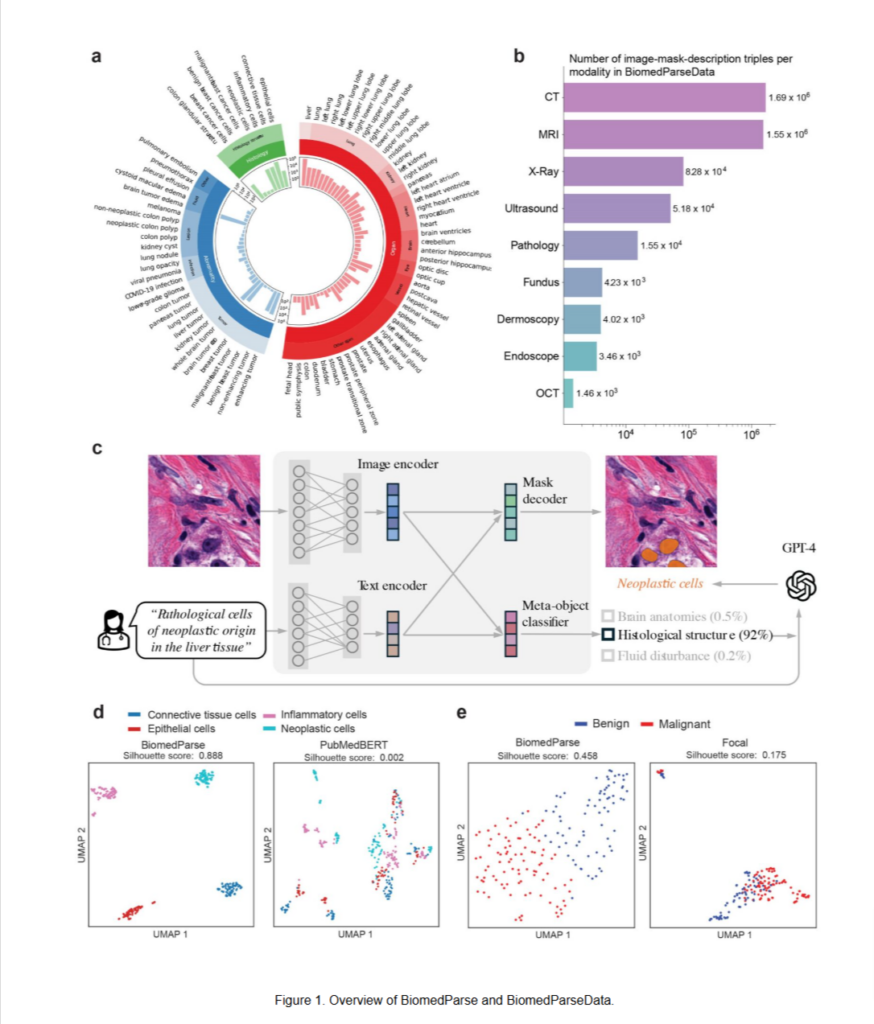
BiomedParse的一个显著优势在于其数据集“BiomedParseData”的开发,这是一个涵盖识别、检测和分割三项任务的大型数据集。数据集的构建是通过利用OpenAI的GPT-4模型,对现有的45个生物医学分割数据集进行大规模的数据合成而实现的。这些数据集中包含了超过600万组图像、分割掩膜和文本描述,通过GPT-4将这些数据进行整合和扩展,形成一个统一的生物医学对象分类体系。
具体来说,GPT-4的使用为数据集的建设提供了强大的支持。首先,它对现有数据集中已经存在的自然语言描述进行了组织和分类,将这些描述与建立的生物医学对象分类体系进行协调一致。然后,GPT-4还用于合成更多对象描述的变化,以便增强模型在文本提示下的鲁棒性。这使得BiomedParse可以更好地应对各种复杂的生物医学场景,提高了其在多样化任务中的表现能力。
三、模型特点与技术优势
BiomedParse在技术上的主要创新之处在于其对三大任务——对象识别、检测和分割——的统一处理,这使得它能够在分析图像时显著减少用户的操作复杂性,同时提高分析结果的准确性和一致性。
1. 全图像解析框架
BiomedParse借鉴了2005年首次提出的“图像解析(Image Parsing)”概念,采用联合学习的方式对对象进行识别、检测和分割。传统的“图像解析”概念基于贝叶斯网络,是一种将多个图像分析任务统一为一个整体的早期尝试,尽管在应用范围和分析能力上存在局限。然而,得益于现代生成式人工智能的快速发展,BiomedParse通过生成式AI技术将这一概念发扬光大,实现了对象之间的联合推理和语义分析。
在使用上,用户只需要输入对某个对象的自然语言描述,模型就能够预测该对象的标签并生成其分割掩膜,无需用户提供对象的具体边界框。这种方式不仅简化了使用流程,还通过联合学习的方法使得对象的检测、识别和分割互相促进,从而提高了整体的图像理解能力。
2. 对复杂和不规则对象的处理
生物医学图像中,常常存在具有复杂和不规则形状的对象,这对传统的分割算法提出了巨大的挑战。即便是使用最先进的对象检测器来提供边界框,对于这些不规则对象的分割仍然困难重重。而BiomedParse通过对象识别、检测和分割的联合学习,学会了如何更好地建模这些复杂形状的对象,其分割性能在面对这些最具挑战性的场景时表现尤为突出。
BiomedParse通过对九种不同的医学影像模态进行大规模训练,涵盖了64种主要对象类型及82种细分类型,能够提供更真实和多样化的生物医学对象的表达。这使得BiomedParse在临床应用中具有更高的适应性和准确性,特别是在需要处理大量不规则对象的情况下。
3. 性能对比与实验评估
在性能评估方面,BiomedParse在大规模测试数据集上表现卓越,涵盖了102,855组图像-掩膜-标签组合。在这些评估中,BiomedParse的表现显著优于其他最佳方法,包括MedSAM、SAM,以及其他如SegVol、Swin UNETR、nnU-Net等方法。在使用最新的对象检测器(如Grounding DINO)来提供边界框时,BiomedParse仍然大幅领先其对手,dice分数在绝对值上超出75到85个百分点。
在具体的实验对比中,BiomedParse不仅在需要提供对象边界框的情况下表现优异,更是在不提供边界框的情况下表现出色,这对于处理真实世界中复杂的生物医学场景具有重要意义。
四、未来发展与应用前景
BiomedParse为生物医学图像的全方位解析提供了坚实的基础,未来的扩展潜力巨大。首先,BiomedParse可以进一步扩展到更多的影像模态和对象类型,使其在更广泛的医学影像中应用。此外,它可以被集成到更高级的多模态框架中,例如LLaVA-Med,这一框架可以通过“与数据对话”的方式实现对图像的交互式分析,进而增强对图像的理解和推理能力。
为了促进生物医学图像分析的研究,BiomedParse已经被开源,并且可以通过Apache 2.0许可在Azure AI平台上进行直接部署和实时推理。这为广大研究者和开发者提供了方便的使用途径,推动了生物医学影像分析领域的进一步发展。未来,BiomedParse可以在精准医疗领域发挥重要作用,例如早期疾病检测、治疗方案制定支持和病情进展监测等,真正实现医学图像分析的智能化和自动化。
五、总结
BiomedParse作为一种基础模型,为生物医学图像分析提供了一种全新的解决方案。通过将对象识别、检测和分割统一到一个框架中,BiomedParse显著提高了图像解析的深度和准确性。这种联合学习的方式不仅减少了用户的手动操作,还通过更为智能的分析提供了更为全面的临床洞察。在面对复杂和多样化的生物医学图像时,BiomedParse展示了其强大的适应性和灵活性。未来,随着更多影像模态和对象类型的纳入,BiomedParse将继续在生物医学图像分析领域发挥核心作用,推动精准医疗的发展与应用。
BiomedParse on GitHub: https://github.com/microsoft/BiomedParse
论文Paper on Nature: A foundation model for joint segmentation, detection and recognition of biomedical objects across nine modalities
数据集BiomedParseData on Huggingface: https://huggingface.co/datasets/microsoft/BiomedParseData