高维临床数据(HDCD)在生物银行(Biobank)级别的数据集中越来越多,但在遗传学研究中的应用仍具有挑战性。论文Unsupervised representation learning on high-dimensional clinical data improves genomic discovery and prediction提出了一种无监督深度学习模型,称为低维嵌入的遗传发现表示学习(REGLE),用于发现遗传变异与HDCD之间的关联。REGLE利用变分自编码器(VAE)计算HDCD的非线性解缠嵌入,这些嵌入成为全基因组关联研究(GWAS)的输入。
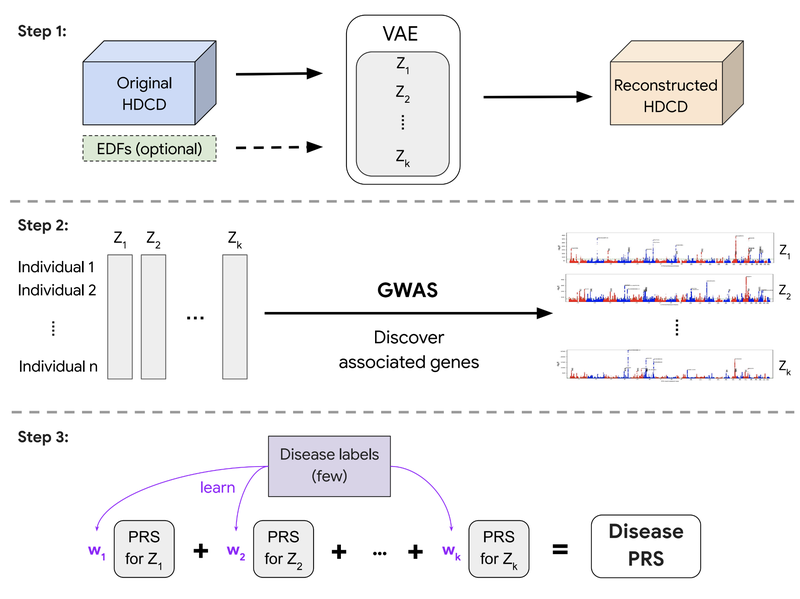
我的初浅理解:通过VAE把高维临床数据(HDCD)压缩到较低维度潜在空间,然后基于基因关联和专家定义特征(Expert-defined Features, EDFs),再对潜在空间的数据进行干涉和调整。🤔
My basic understanding is: High-dimensional clinical data (HDCD) is compressed into a lower-dimensional latent space using a Variational Autoencoder (VAE). Then, based on gene associations and expert-defined features (EDFs), the data in the latent space is further manipulated and adjusted.
该研究由如下人员合作完成:Justin Cosentino, Babak Behsaz, Yuchen Zhou, Zachary R. McCaw, Howard Yang, Andrew Carroll, Cory Y. McLean (Google), Davin Hill (Northeastern University), Tae-Hwi Schwantes-An, Dongbing Lai (Indiana University), John Bates (Verily), Brian D. Hobbs, Michael H. Cho (Brigham and Women’s Hospital & Harvard Medical School), Robert Luben, Anthony P. Khawaja (Moorfields Eye Hospital & University College London).
以下为论文内容概要(ChatGPT 4o生成):
一、方法
- 模型架构:
- VAE训练: 使用变分自编码器(VAE)对HDCD进行压缩和重构,生成低维的非线性嵌入。
- GWAS: 在每个嵌入坐标上独立进行GWAS。
- PRS构建: 使用嵌入坐标的多基因风险评分(PRS)作为一般生物功能的遗传评分,结合它们以创建特定疾病或特征的PRS。
- 数据集:
- 使用UK Biobank中的呼吸和循环HDCD数据,分别为肺功能测量的呼吸图和血液体积变化的光电容积图(PPG)。
二、结果
- 嵌入生成与解释:
- 呼吸图(SPINCs)和残差呼吸图(RSPINCs): 训练卷积VAE生成呼吸图嵌入。通过注入五个专家定义特征EDFs(FEV1、FVC、FEV1/FVC、PEF和FEF25-75%)作为解码器的输入来生成RSPINCs。
- PPG嵌入(PLENCs)和残差PPG嵌入(RPLENCs): 训练卷积VAE生成PPG嵌入,通过注入五个PPG EDFs生成RPLENCs。
- GWAS分析:
- 呼吸功能: SPINCs和RSPINCs分别检测到575和659个独立的全基因组显著性(GWS)位点,其中许多是以前未检测到的。
- 循环功能: PLENCs和RPLENCs分别检测到90和75个独立的GWS位点,其中大部分为新发现。
- PRS构建与评估:
- 肺功能相关疾病(哮喘和COPD): SPINCs和EDFs+RSPINCs的PRS在UK Biobank中显著改善了哮喘和COPD的预测。
- 心血管功能相关疾病(高血压和SBP): PLENCs和RPLENCs的PRS在多个独立数据集(如COPDGene、eMERGE III和EPIC-Norfolk)中改善了高血压和SBP的预测。
三、讨论
- 模型优势:
- 无监督学习: REGLE无需标签,能有效利用HDCD进行遗传发现。
- 低维非线性嵌入: VAE生成的嵌入能更好地捕捉HDCD中的遗传信号。
- 模型局限性:
- GWAS优化: 当前方法较保守,可进一步优化以合并多个嵌入坐标的信号。
- 模型泛化性: 模型仅在UK Biobank上训练,需在其他数据集上验证其泛化性。
- 未来研究方向:
- 集成多种HDCD: 将其他类型的HDCD(如影像数据)纳入模型,进一步揭示复杂疾病的遗传机制。
- 优化嵌入目标: 引入最大化遗传力的目标函数,提升嵌入的遗传分析性能。
四、结论
REGLE通过生成HDCD的低维非线性嵌入,显著改善了遗传发现和疾病预测,为高维临床数据在遗传学研究中的应用提供了新方法和新视角。
五、主要贡献
- 提出了无监督学习模型REGLE,有效利用高维临床数据进行遗传发现。
- 通过实验证明REGLE在UK Biobank数据上对多种疾病的预测能力优于传统方法。
- 提供了详细的模型架构和评估方法,为后续研究提供了参考。