根据世界卫生组织(WHO)的数据,每年全球有超过125万人因对抗生素等药物免疫的感染而死亡。到2050年,这一数字可能会增加到1000万人。而在未来六年内,约2400万人可能会因为治疗传染病的高昂费用而陷入极端贫困。
使用人工智能的研究人员正在挖掘早已灭绝的物种,如猛犸象和巨型树懒的DNA,希望找到古代基因组的秘密,以帮助对抗当今最具感染性的病原体。宾夕法尼亚大学(University of Pennsylvania)的教授Cesar de la Fuente博士领导着一个研究团队,他们利用人工智能来探测灭绝的史前生物的DNA,寻找对抗危险的耐药性微生物的新解决方案。这种技术被de la Fuente博士及其团队称为“分子复生(Molecular De-extinction)”,并在2024年6月发表在《自然生物医学工程》期刊上的一篇论文(Deep-learning-enabled antibiotic discovery through molecular de-extinction)中进行了详细描述。
该研究解决了抗生素耐药性这一关键问题。作者提出了一种通过深度学习方法探索已灭绝生物蛋白质组(称为“灭绝组”)以发现具有潜在抗菌特性的肽序列的创新方法,这些肽可能有助于对抗耐药性病原体。
论文作者为Fangping Wan, Marcelo D. T. Torres, Jacqueline Peng和Cesar de la Fuente-Nunez,均来自宾夕法尼亚大学。
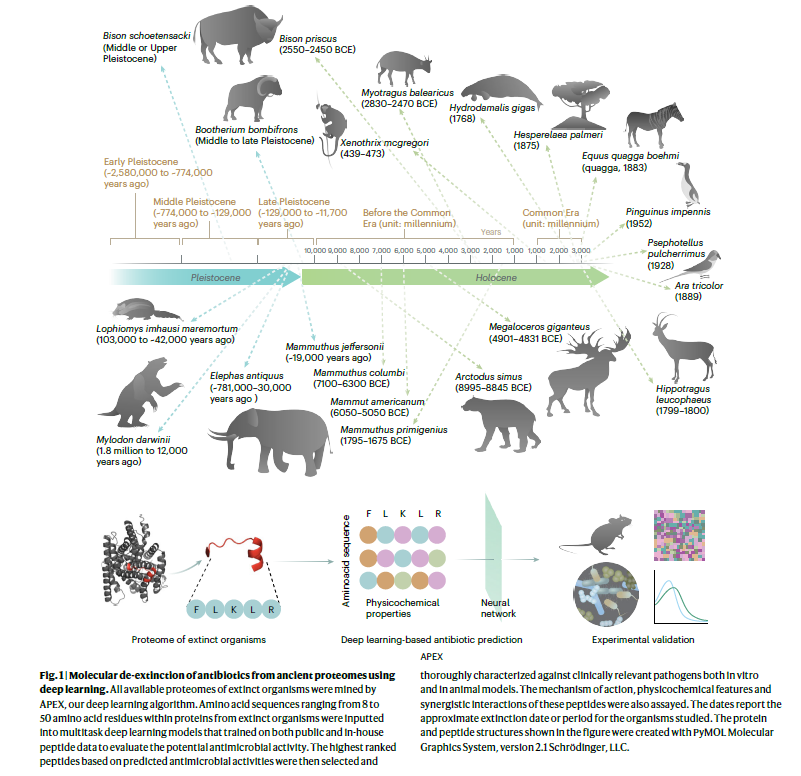
以下为论文内容概述:
一、方法论
1. 分子复生:
- 分子复生的概念涉及从已灭绝生物中复活分子,以应对当前的生物医学挑战。
- 作者利用深度学习技术,挖掘已灭绝生物的蛋白质组,寻找具有潜在抗菌活性的序列。
2. 深度学习模型 – APEX:
- 作者开发了一种名为APEX的深度学习模型,这是一种多任务学习架构,旨在预测肽的抗菌活性。
- APEX使用来自内部数据集和公共的抗菌肽活性和结构数据库(DBAASP)的数据进行训练。
- 该模型使用结合了递归和注意力机制的编码器神经网络,从肽序列中提取隐藏特征,然后将这些特征输入到两个全连接神经网络(FCNN)中,以预测抗菌活性。
3. 灭绝组的挖掘:
- 研究收集了208个已灭绝物种的12,860个蛋白质序列。经过筛选,剩下了5,190个蛋白质,最终识别出10,311,899个肽序列。
- 使用APEX预测这些序列的抗菌活性,结果发现了37,176个肽被预测为具有广谱抗菌活性,其中11,035个肽在现存生物中未发现。
二、实验验证
作者合成了69个APEX预测的肽,并对其抗菌活性进行了实验验证。关键发现包括:
1. 抗菌活性:
- 这些肽表现出不同水平的抗菌活性,命中率为59%,即识别出具有活性肽的比例。
- 研究重点介绍了几种具有显著抗菌特性的肽,包括来自猛犸象的mammuthusin-2和来自古代海牛的hydrodamin-1。
2. 作用机制:
- 大多数肽通过使细胞质膜去极化来杀死细菌,这一机制不同于已知的抗菌肽,它们通常靶向外膜。
- 研究还探讨了这些肽的理化性质,指出它们在氨基酸组成和二级结构方面与现代抗菌肽存在差异。
三、主要结果与意义
研究表明,深度学习可以有效地挖掘已灭绝生物的蛋白质组,从而发现新的抗生素候选者。这些肽的发现为开发可能对抗抗生素耐药性细菌的新型抗生素开辟了新的途径。