论文Genome modeling and design across all domains of life with Evo 2介绍了Evo 2,一个训练于涵盖所有生命领域的基因组数据集的生物学基础模型,该模型可用于深入理解和设计生命中的基因组功能。Evo 2在9.3万亿个DNA碱基对数据上训练,并能够以单碱基分辨率预测遗传变异的功能影响。
论文作者和所属机构如下:

一、引言
生物学研究的核心之一是揭示DNA中信息的编码方式,DNA在所有生命形式中扮演着至关重要的角色。随着基因组学工具的发展,基因测序、基因合成以及基因编辑技术为生物学研究带来了巨大突破。然而,尽管这些技术能够帮助我们理解DNA序列和生物学功能之间的关系,要设计新的生物系统,仍然需要更深入的理解基因组的复杂性。为此,研究人员提出了Evo 2模型,它是一个生物学基础模型,通过在包含9.3万亿DNA碱基对的训练集上进行学习,能够准确地预测遗传变异对基因组功能的影响。Evo 2在不进行特定任务微调的情况下,可以通过单纯分析DNA序列,预测变异的功能效应,包括非编码突变和临床上有重要意义的BRCA1突变。
二、Evo 2模型架构与训练过程
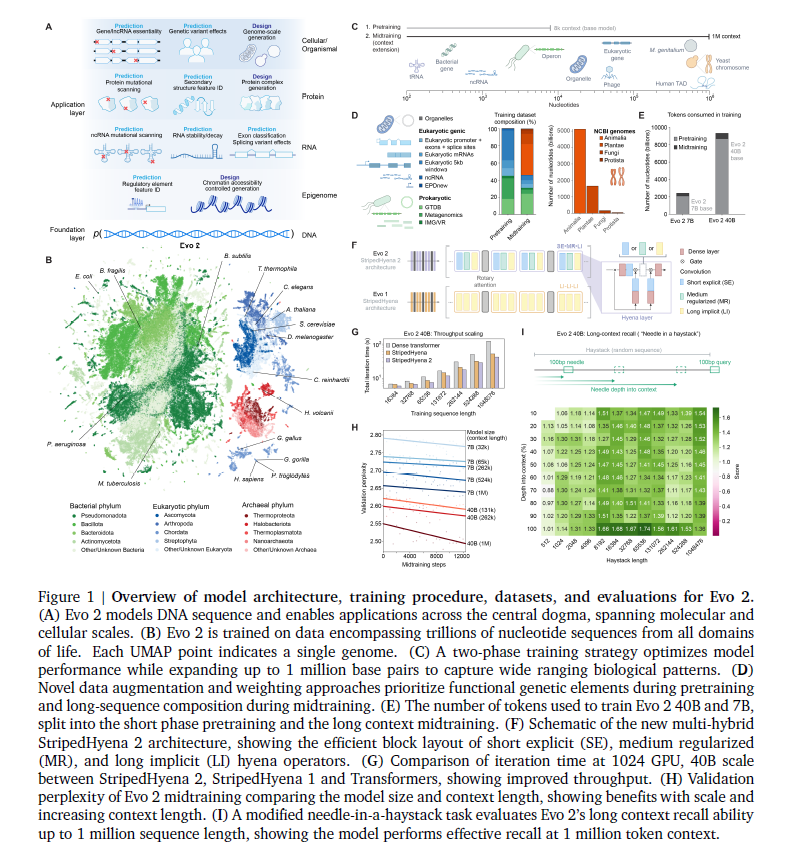
Evo 2采用了一种多混合卷积架构(StripedHyena 2),这是第一个基于输入依赖卷积操作符的多混合架构。该架构通过结合三种不同类型的卷积运算符(短、长和中等长度的卷积),显著提高了大规模训练的效率,使得Evo 2能够处理长达100万碱基对的DNA序列。相比传统的Transformer架构,Evo 2在大规模训练中的速度更快,处理更长序列时表现更加高效。
Evo 2在OpenGenome2数据集上进行训练,该数据集包含来自细菌、古菌、真核生物和病毒的基因组序列,数据的总量达到了9.3万亿个token。训练过程分为两个阶段:第一阶段是预训练,使用较短的上下文窗口(8,192个token),重点学习功能性基因元素;第二阶段是中期训练,将上下文窗口扩展到1百万个token,以学习基因组序列中各个元素之间的长距离关系。Evo 2的训练数据没有包含感染真核生物的病毒基因组,这一数据选择有助于避免模型在该领域的语言建模能力偏弱,确保其在其他领域的有效性。
三、Evo 2的预测与生成能力
Evo 2的能力不仅仅局限于基因突变的预测,它还能够生成新的基因组序列,并在多个生物学任务中表现出色。Evo 2通过“零样本(zeor-shot)”学习,无需进行特定任务的微调,即可对DNA、RNA和蛋白质突变的影响进行预测。在“零样本(zeor-shot)”任务中,Evo 2表现出色,能够预测突变对分子功能和有机体适应性的影响,尤其在非编码区域和剪接变异的预测上表现突出。研究人员还发现,Evo 2能够预测各种突变类型,包括非同义突变、错义突变、插入或缺失(indels)等,对其潜在致病性、剪接效应及生物学功能进行精准评分。
Evo 2在基因组序列生成方面也表现出色。该模型可以生成完整的基因组序列,包括线粒体基因组、最小细菌基因组和酵母染色体等,生成的序列具有比以前模型更高的自然性和连贯性。这意味着Evo 2不仅能够生成符合生物学特性的基因序列,还能够生成具有生物学功能的序列。这一能力为合成生物学提供了重要工具,能够用于创建新的生物体或调节现有生物体的基因组。
四、机制可解释性与特征分析
Evo 2通过先进的机制可解释性方法,揭示了其内部表示的生物学特征。研究团队利用稀疏自编码器(SAEs)对Evo 2的神经元激活模式进行解码,从而发现了一些重要的生物学特征。通过这些特征分析,研究人员识别出了与外显子-内含子边界、转录因子结合位点、蛋白质结构元素、噬菌体基因组区域等密切相关的特征。这些特征不仅帮助我们理解Evo 2是如何学习并处理基因组数据的,还可以应用于基因组学的发现任务,例如预测和识别新的遗传元件或表型特征。
例如,Evo 2在学习基因组数据时,能够自动识别并激活与噬菌体相关的特征,这些特征与噬菌体区域以及CRISPR阵列中的间隔序列有关。类似地,Evo 2还能够识别与蛋白质二级结构(如α螺旋和β折叠)相关的特征,进一步证明了基因组语言模型能够超越DNA序列本身,理解生物学结构信息。
五、表观基因组生成与推理引导
Evo 2不仅可以生成DNA序列,还能够通过推理引导技术生成表观基因组结构。表观基因组的调控涉及DNA的化学修饰,如DNA甲基化和组蛋白修饰,这些修饰对基因表达具有重要影响。在这项研究中,Evo 2被用来生成具有特定染色质可及性模式的DNA序列。染色质可及性是指DNA是否能被转录机械所接触到,从而影响基因的表达。通过结合外部的表观基因组预测模型(如Enformer和Borzoi),Evo 2能够在生成DNA序列的同时,优化这些序列的染色质可及性,使其符合预设的模式。
研究团队使用了一个“束搜索”算法,通过对部分生成的DNA序列进行评估,选择最符合期望染色质模式的序列。这个方法不仅能生成特定区域的染色质开放区域,还可以控制染色质闭合区域的长度和位置,最终生成具有特定功能的表观基因组。
六、讨论
Evo 2展示了基因组语言模型在生物学领域的广泛应用潜力。其通过在超大规模的基因组数据集上训练,能够在分子、细胞以及有机体层面预测基因突变的影响,尤其在非编码区域的预测方面,Evo 2超越了现有的其他基因组语言模型。Evo 2不仅在“零样本(zeor-shot)”变异效应预测中取得了显著进展,还能够生成具有生物学功能和自然性的全基因组序列。此外,Evo 2在推理时进行的表观基因组设计,展示了它在复杂生物学特性设计中的巨大潜力。
七、结论
Evo 2是一个强大的生物学基础模型,它在预测基因变异效应、生成基因组序列以及设计表观基因组等任务中表现出了卓越的能力。Evo 2的发布不仅为基因组学研究提供了新的工具,也为合成生物学、个性化医疗和基因组设计等领域提供了重要支持。未来,Evo 2将结合更多生物学数据和计算技术,推动基因组学和生物学设计的智能化发展,帮助研究人员解锁生命的复杂性。