论文Delineating the effective use of self-supervised learning in single-cell genomics详细探讨了自监督学习(Self-Supervised Learning,SSL)在单细胞基因组学(Single-Cell Genomics,SCG)中的有效性与应用,重点分析了如何通过SSL从大规模、未标注的数据中提取有意义的表示。研究为SCG提供了新的一种方法框架,并通过大量实证分析,比较了不同的SSL策略在多个下游任务中的表现。
论文作者为Till Richter, Mojtaba Bahrami, Yufan Xia, David S. Fischer & Fabian J. Theis,来自Institute of Computational Biology, Technical University of Munich, Eric and Wendy Schmidt Center at the Broad Institute.
1. 研究背景与挑战
单细胞基因组学领域迅速发展,主要得益于单细胞RNA测序技术的进步。随着单细胞数据集的规模和复杂性增加,SCG面临着许多挑战:
- 数据稀疏性和异质性:单细胞RNA测序数据通常会受到技术批次效应的影响,并且基因表达数据存在较大的噪声。
- 标签质量问题:由于手动注释单细胞类型涉及高成本且不易获得高质量的标签,很多情况下无法获得足够的标注数据。
- 大规模数据处理:随着数据集规模的扩大,如何处理包含数百万细胞的大规模数据集成为一大难题。
传统的监督学习方法通常依赖于大规模的标注数据集,但标注数据的获取是单细胞基因组学中一个瓶颈。自监督学习(SSL)则通过学习数据内在的结构,而不依赖人工标签,能够有效地解决这一问题。
2. 研究目标
论文的核心目标是探讨自监督学习在单细胞基因组学中的应用,特别是它如何通过无标签数据来增强不同下游任务的性能。具体目标包括:
- 确定自监督学习在单细胞基因组学中的应用场景。
- 基准测试不同的SSL策略(如遮掩自动编码器和对比学习),并比较其在多种任务中的表现。
- 确定最有效的预训练任务(pretext task),为实际应用提供方法论指导。
3. 研究方法与框架
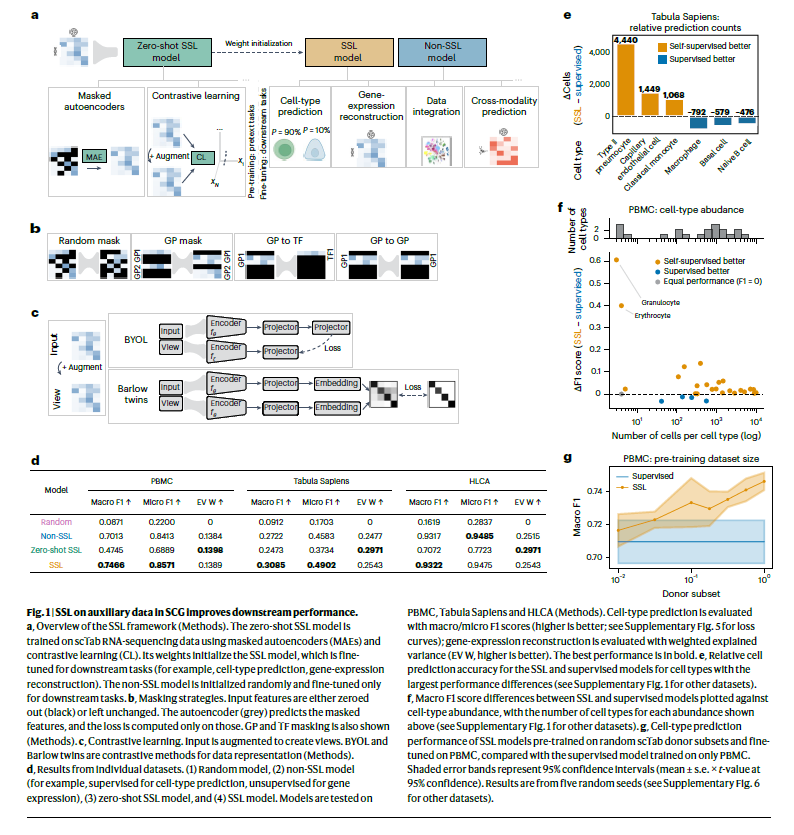
论文提出了一个基于全连接网络(Fully Connected Networks,FCNs)的SSL框架,结合遮掩自动编码器(Masked Autoencoders,MAE)和对比学习(Contrastive Learning)两种自监督方法,来探索其在SCG中的应用。
3.1 自监督学习框架
该框架分为两个阶段:
- 预训练阶段(Pretraining):模型在没有标签的情况下学习数据的潜在结构。使用的方法包括遮掩自动编码器(MAE)和对比学习(CL)。在这一阶段,模型通过自监督任务(如遮掩部分输入特征并重构这些特征)来学习数据的表示。
- 微调阶段(Fine-tuning):将预训练模型应用于具体的下游任务,如细胞类型预测、基因表达重建等。微调阶段通常会在标注数据上进行,以提高模型在具体任务上的表现。
3.2 SSL任务设计
- 遮掩自动编码器(Masked Autoencoders,MAE):该方法通过随机遮掩输入特征(如基因表达数据中的基因值),并要求模型重建这些遮掩的部分。论文使用了多种遮掩策略,包括:
- 随机遮掩:随机选择50%的基因进行遮掩,这种方法偏向于减少偏差。
- 基因程序遮掩(GP Masking):根据基因的已知功能遮掩一组特定的基因,以加强模型对生物学关系的理解。
- 转录因子(TF)遮掩:通过仅遮掩特定基因程序(GP)与转录因子之间的关系来训练模型,进一步细化生物学结构。
- 对比学习(Contrastive Learning,CL):通过对比样本间的相似性和差异性来训练模型。论文使用了两种对比学习策略:
- BYOL(Bootstrap Your Own Latent):通过教师-学生网络架构来优化模型,避免表示崩溃(即网络输出恒定值的问题)。
- Barlow Twins:通过最大化信息内容来避免表示崩溃,特别适用于处理高维度、复杂数据。
4. 主要发现与实验结果
4.1 自监督学习在小数据集上的表现
论文首先测试了SSL模型在小规模数据集上的表现,包括人类肺细胞图谱(HLCA)、外周血单核细胞(PBMC)和Tabula Sapiens数据集。通过与传统监督学习方法进行比较,发现自监督预训练显著提高了细胞类型预测的准确性:
- 在PBMC数据集上,宏观F1分数从0.7013提升至0.7466。
- 在Tabula Sapiens数据集上,宏观F1分数从0.2722提高到0.3085。
这种提升尤其在细胞类型分类的平衡性较差的任务中显得尤为明显。
4.2 零样本学习的优势
在零样本学习(zero-shot learning)设置下,即仅通过自监督预训练得到的模型而不进行微调,SSL模型展示了强大的零样本性能。在scTab测试集上的宏观F1分数达到了0.6725,表现接近经过微调的监督学习模型。对于未见数据集(如人脑图谱和大猩猩数据集),SSL模型的性能同样优于无监督模型。
4.3 SSL在跨模态预测中的表现
为了评估SSL是否能在多模态数据中提升性能,论文使用了NeurIPS多组学数据集,该数据集包含了RNA-seq与蛋白质组学数据。通过SSL预训练,模型在RNA-seq数据的基础上预测蛋白质计数,结果表明,SSL显著提高了预测精度,相比于基线方法,Pearson相关系数从0.8809提升至0.8943。
4.4 数据集成中的优势
在数据集成任务中,SSL预训练有效地消除了不同数据集之间的批次效应,保证了生物学变异性的保留。通过scIB数据集成基准评估,SSL模型在批次校正和生物学保留方面优于传统无监督方法。特别是在使用少量数据集时,SSL模型表现出了显著的优势。
5. 讨论与结论
论文总结了自监督学习在单细胞基因组学中的应用场景,并针对不同任务提出了实用的建议。研究表明,SSL在以下几种场景下尤其有用:
- 迁移学习:通过预训练模型在大规模辅助数据集上,SSL能够提升在小规模数据集上的学习效果。
- 跨模态数据预测:SSL能够有效地利用一种模态的数据来提升另一种模态的预测能力。
- 数据集成:SSL在处理不同来源的数据时表现出色,尤其在批次效应校正和数据融合方面具有优势。
然而,研究也表明,SSL并非适用于所有情况。在同一数据集上进行预训练和微调时,SSL的表现与传统的监督学习方法相当,且在某些任务中,对比学习方法的效果不如遮掩自动编码器。
6. 未来研究方向
- 模型架构扩展:未来研究可以探索其他架构(如Transformer)与SSL结合的效果。
- 稀有数据场景:SSL在处理小规模、稀有数据集(如时序数据或动态建模)中的潜力值得进一步挖掘。
- SSL预训练策略的优化:不同预训练任务的选择可能对任务的表现产生重大影响,未来可以进一步优化任务设计,提升SSL的泛化能力。