论文Boltz-1: Democratizing Biomolecular Interaction Modeling(《Boltz-1: 民主化生物分子相互作用建模》)介绍了一种名为Boltz-1的开源深度学习模型,用于预测生物分子复合物的三维结构。Boltz-1作为首个达到AlphaFold3精度并完全开源的模型,通过架构创新、数据优化和推理流程的改进,显著降低了生物分子建模的门槛,使其能够被更广泛的研究群体使用。Boltz-1的开放源码、模型权重及数据集,为生物信息学和结构生物学的研究提供了强大的工具,极大地促进了生物分子相互作用的研究。
Boltz-1的发布标志着生物分子建模工具的民主化,未来研究人员可以基于Boltz-1进行进一步的改进和创新,加速药物设计和结构生物学领域的突破,推动对生物分子相互作用的深入理解。
论文作者为Jeremy Wohlwend, Gabriele Corso, Saro Passaro, Mateo Reveiz, Ken Leidal, Wojtek Swiderski, Tally Portnoi, Itamar Chinn, Jacob Silterra, Tommi Jaakkola , Regina Barzilay,来自MIT和Genesis Therapeutics。
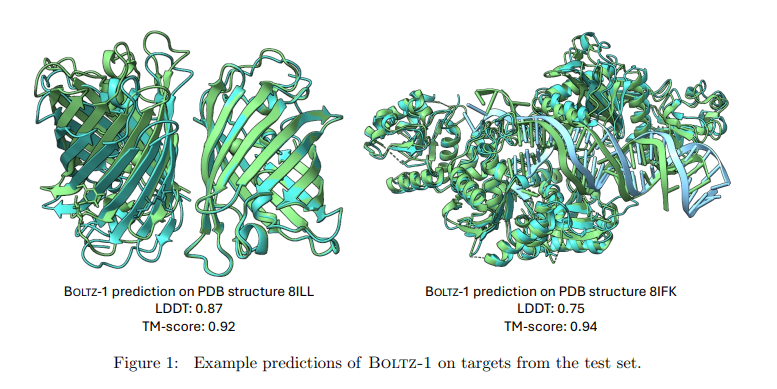
以下为论文概要内容介绍:
1. 引言与背景
Boltz-1的研究背景涉及生物分子相互作用在药物发现、蛋白质设计等领域的重要性。传统上,预测生物分子复合物的三维结构是一项复杂且计算密集的任务。AlphaFold2开启了单链蛋白质结构预测的新时代,而后续的AlphaFold3在预测任意生物分子复合物的精度上达到了新的高度。然而,AlphaFold3的闭源特性以及其对硬件资源的高需求,限制了其在学术界和工业界的广泛应用。因此,Boltz-1的设计初衷是通过开源和架构优化,使得这种高精度的结构预测工具能够被更多的研究人员和机构所使用,以推动全球生物分子建模的进步。
Boltz-1不仅延续了AlphaFold系列的基本架构,还在模型效率、数据处理、推理精度等方面做出了诸多创新。论文中明确指出,Boltz-1的目标是“民主化”生物分子建模,即使有限资源的团队也能利用这一强大的工具。这种开放性和广泛可访问性为生物信息学和结构生物学的研究提供了新的可能。
2. 数据处理管道
2.1 数据来源与处理
Boltz-1在训练中使用了截至2021年9月30日发布的所有PDB结构,分辨率至少为9Å。这与AlphaFold3相同,确保了数据的一致性,使得两者之间的性能比较具有可比性。数据来源包括蛋白质数据银行(PDB),其中每个多聚体链的参考序列通过对齐算法对残基进行了匹配,并使用了RDKit的ETKDGv3算法来生成小分子的构象。
Boltz-1在输入数据中不包含模板数据,认为这些模板对大型模型的影响微乎其微。相反,Boltz-1通过多序列比对(MSA)、小分子的笑表达式(SMILES)以及基因组序列来表示蛋白质、配体和核酸,这些输入被进一步用于结构预测。
2.2 验证和测试集的策划
Boltz-1采用与AlphaFold3类似的验证和测试集划分策略,通过MMseqs的easy-cluster命令根据序列同一性对蛋白质序列进行聚类。为了避免数据泄露,所有训练集中的序列被排除在验证和测试集之外。最终,训练集、验证集和测试集分别涵盖了1728个、553个和593个结构。这种严格的数据划分确保了模型在广泛生物分子上的泛化能力。
2.3 密集的MSA配对算法
为了捕捉多聚体蛋白质复合物中进化共生的氨基酸对,Boltz-1提出了一种改进的MSA配对算法,该算法利用分类学信息保持MSA的密度,同时在配对信号和序列冗余之间寻找平衡。这种改进的配对方法帮助模型更好地处理蛋白质-蛋白质相互作用预测的挑战。
2.4 统一裁剪算法
Boltz-1引入了一种新的统一裁剪算法,通过在空间裁剪和连续裁剪之间进行插值,使得模型在训练时能接收到多样化的裁剪策略信号。在具体实现上,裁剪算法通过定义邻域(neighborhood)来实现,这些邻域围绕特定的中心标记增量地添加到裁剪区域。该算法确保了每次训练样本中的结构裁剪都具有一定的随机性和多样性,从而提高模型的泛化能力。
2.5 结合位点的条件化
Boltz-1提出了一种稳健的结合位点条件化方法,以解决实际应用中用户对结合位点了解不足的问题。与AlphaFold3的设计不同,Boltz-1保留了单一的统一模型,在训练过程中有30%的迭代中随机选择结合位点信息。该信息通过几何分布选择一部分结合位点残基,然后将其编码为额外的特征,这使得模型在推理时可以更好地处理部分结合位点已知的情况。
3. 模型架构与训练
3.1 模型架构修改
Boltz-1的模型架构建立在AlphaFold3的基础上,但在以下方面做了重要改进:
- MSA模块重排:原本的AlphaFold3架构中,单序列和序列对的表示是按固定顺序更新的,Boltz-1则调整了这一顺序,使得MSA的单序列更新可以更快地传递到序列对的表示中。
- 变压器层的调整:AlphaFold3中的变压器层在进行注意力偏置和条件转换块操作时,没有使用残差连接,这使得反向传播可能更加困难。Boltz-1改进了这一点,增加了残差连接,使得模型在深层次的训练过程中能够保持梯度的有效传播。
3.2 Kabsch扩散插值
在训练过程中,Boltz-1采用了基于Kabsch算法的刚性对齐插值方法。AlphaFold3使用刚性对齐来减少去噪损失的变化,但这种对齐本身可能会导致推理阶段的误差积累。Boltz-1通过在每一步的扩散插值后进行刚性对齐,保证插值结构比噪声结构更接近去噪样本,从而提高了模型的稳定性。
3.3 置信度模型
Boltz-1对置信度模型的设计与AlphaFold3不同。Boltz-1的置信度模型不再只是四个PairFormer层,而是包含了模型主干的所有组件,并且以训练好的主干权重进行初始化。模型在推理过程中集成了每一步扩散后的表示,通过时间条件化的递归块聚合这些表示,以提高预测的可靠性。
3.4 模型优化
Boltz-1在以下几个方面进行了模型优化以提高计算效率:
- 序列局部原子表示:Boltz-1对序列局部原子的注意力计算进行了预处理,使得注意力操作可以在稀疏的块中并行执行,从而显著提升了计算效率。
- 注意力偏置共享与缓存:去噪模型中的注意力偏置和中间表示并不依赖于具体的输入结构或扩散时间,因此Boltz-1将这些计算提前并在所有扩散步骤中共享,从而大幅降低了计算开销。
4. 结果与评估
Boltz-1在多样化的测试集和CASP15基准数据集上进行了全面评估,结果表明:
- 整体性能:Boltz-1在测试集和CASP15数据集上的中位LDDT和中位TM评分与Chai-1相当,这表明模型在预测通用生物分子结构方面具备了良好的精度。
- 蛋白质-蛋白质相互作用:在蛋白质-蛋白质相互作用预测中,Boltz-1在CASP15数据集上的表现优于Chai-1。
- 蛋白质-配体相互作用:Boltz-1在CASP15中表现出显著优势,尤其是在配体结合位点的预测中,显示了较强的泛化能力。
这些结果展示了Boltz-1在生物分子结构预测的多个重要指标上达到了当前最先进的水平。
5. 局限性
尽管Boltz-1在多项基准测试中表现出色,但模型在一些复杂结构预测中出现了幻觉(hallucination)问题,主要表现为相同的分子链被预测为完全重叠。这种现象可能由以下原因引起:
- 数据中的重叠链和配体:训练集中存在部分重叠的配体,可能为模型的学习带来了误导性信号。
- 训练裁剪尺寸不足:由于计算资源的限制,模型在训练中使用的裁剪尺寸相对较小,难以有效捕捉大型复杂结构的全局上下文信息。
Boltz-1:https://jclinic.mit.edu/boltz-1/
Boltz-1 on GitHub:https://github.com/jwohlwend/boltz
Genesis Therapeutics公司介绍:
Genesis Therapeutics是一家位于美国加利福尼亚州的人工智能(AI)制药初创公司,成立于2019年。公司致力于将AI技术与物理和化学知识相结合,设计和开发新型小分子候选药物,以治疗严重和衰弱性疾病。
Genesis Therapeutics的目标是通过其先进的AI平台,解决传统药物发现方法难以攻克的靶点,开发出高效、高选择性和生物利用度的下一代疗法,为患者带来新的希望。
Genesis Therapeutics开发了名为GEMS(Genesis Exploration of Molecular Space)的AI平台。该平台集成了基于深度学习的预测模型、分子模拟和化学感知语言模型,用于加速和优化小分子药物的发现。其中,Dynamic PotentialNet模型通过分子模拟和实验数据进行训练,学习分子结合亲和力背后的物理相互作用,从而基于分子的3D结构进行准确预测。
Genesis Therapeutics与多家大型制药公司建立了合作关系:
- 礼来公司(Eli Lilly): 2022年5月,Genesis与礼来达成合作,利用其AI平台,针对多个治疗领域的多达5个靶点发现创新疗法。根据协议,Genesis将获得2000万美元的预付款,并有资格获得高达6.7亿美元的里程碑付款。
- 吉利德科学(Gilead Sciences): 2024年9月,Genesis与吉利德宣布达成战略合作,共同发现和开发跨多个靶点的新型小分子疗法。此次合作将部署Genesis的GEMS平台,以协助生成和优化吉利德所选靶点的分子。
Genesis Therapeutics由Evan Feinberg博士和Ben Sklaroff联合创立。Feinberg博士在斯坦福大学获得生物物理学博士学位,是PotentialNet算法的发明者。Sklaroff担任公司首席技术官(CTO),在加州大学伯克利分校获得电气工程和计算机科学学位,曾是3D打印公司Markforged的早期成员之一。